Auteur : Paul Passy
Segmentation d’images
La segmentation d’images est une technique de traitement d’images qui permet d’individualiser des zones homogènes sur une image. Ces zones homogènes sont appelées segments. Cette technique permet par exemple de classifier une image non pas en individualisant les pixels un par un mais en replaçant chaque pixel dans son contexte immédiat. L’avantage de cette technique est de fournir des segments d’images, i.e. des zones homogènes d’images, qu’il est possible de transformer en polygones vecteurs. Cette transformation en polygones permet de calculer de nouveaux attributs pour chaque segment comme une superficie, un périmètre, un indice de forme, un indice de voisinage… Cette technique de segmentation est également connue sous l’acronyme anglais OBIA pour Object-Based Image Analysis.
Cette technique de segmentation est plus particulièrement utilisée sur des images en très haute résolution spatiale (THRS), comme les images issues du capteur Pléiades (ou tout autre capteur satellite de THRS) ou les orthophotographies. Cependant rien n’empêche d’appliquer cette technique à tout type d’images quelle que soit sa résolution spatiale.
Le but d’une segmentation est la plupart du temps le calcul d’une occupation du sol ou l’identification de certaines classes ou certains objets. C’est une technique qu’il est, par exemple, possible d’employer pour détecter les espaces verts urbains à partir d’othophotographies de milieux urbains ou bien pour détecter des voitures sur des parkings ou des bateaux dans un port.
Le terme de Segmentation recouvre souvent quatre étapes qui s’enchaînent :
la segmentation proprement dite pour détecter les segments homogènes de l’image
la vectorisation des segments obtenus en transformant les segments en polygones vecteurs
le calcul de primitives pour caractériser ces segments, comme des valeurs de réflectances moyennes, des surfaces, des indices de formes, des moyennes de NDVI, MNDWI,..
la classification supervisée de ces segments polygonisés pour détecter les objets d’intérêt
Différents logiciels de géomatique proposent des fonctionnalités plus ou moins complètes de segmentations d’images. Néanmoins, Orfeo ToolBox (OTB) est un de ceux proposant la solution la plus complète.
Dans la suite de cette section, nous prendrons l’exemple de l’extraction des espaces verts des alentours de la Place d’Italie dans le 13ème arrondissement de Paris, à partir d’une orthophotographie infrarouge centrée sur cette place. L’orthophotographie est issue de la BD Ortho fournie par l’IGN via son portail Géoservices. Sa résolution spatiale est de 20 cm, elle se compose de trois canaux : proche-infrarouge (bande 1), rouge (bande 2) et vert (bande 3) et est référencée en Lambert 93. Une vue en fausses couleurs de la zone prise en exemple est montrée sur la figure suivante (Fig. 395).

Fig. 395 Orthophotograhie centrée sur la Place d’Italie (Paris 13ème), en rouge le canal du proche-infrarouge, en vert le canal du rouge et en bleu le canal du vert.
Note
La segmentation d’images est une technique puissante mais qui s’avère être très procédurière. Il faut bien être attentif aux différentes étapes. Le géomaticien distrait a vite fait de se perdre dans les méandres de cette technique. Mais une fois la logique acquise, le processus coule de source.
Table des matières
Segmentation d’images avec Orfeo ToolBox
Dans OTB il existe plusieurs façons de procéder pour segmenter une image, nous verrons ici une des plus utilisées. Nous commencerons par détailler l’étape de Segmentation proprement dite. Cette étape se subdivise elle-même en trois sous étapes. Dans un premier temps nous verrons comment réaliser ces trois sous étapes une par une puis nous verrons une solution qui permet de les chaîner en une seule sous étape.
Segmentation de l’image en trois sous-étapes
Nous commencerons par voir le processus de segmentation de façon détaillée en passant par les trois (ou quatre) sous-étapes qui sont :
le lissage de l’image initiale
la segmentation de cette image lissée
la fusion des plus petits segments avec les grands segments voisins les plus similaires (facultatif)
la vectorisation de la segmentation
Lissage de l’image initiale
Cette étape de lissage n’est pas indispensable mais elle permet d’accélérer les traitements. Le but est de simplifier l’image initiale en homogénéisant les zones similaires d’un point de vue radiométrique. Le module permettant ce lissage se nomme MeanShiftSmoothing (Fig. 396).
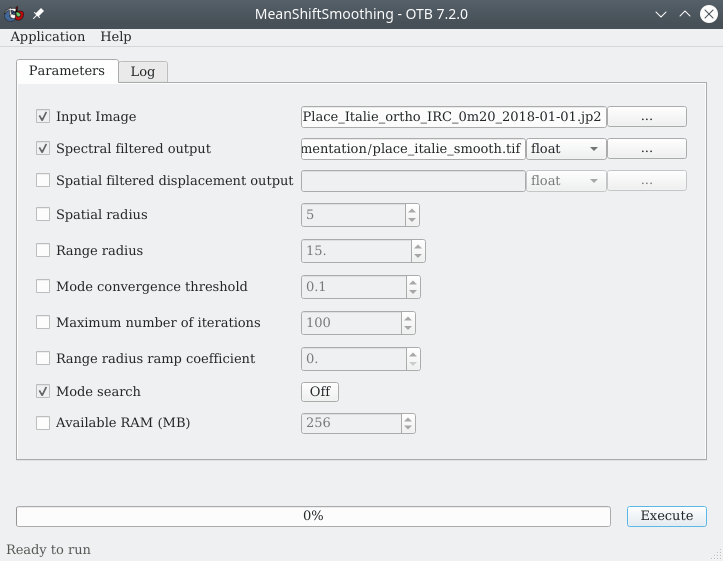
Fig. 396 Lissage de l’image initiale avec le module MeanShiftSmoothing de OTB.
Dans le menu de ce module, à la ligne Input Image nous pointons vers l’image à lisser, ici Place_Italie_ortho_IRC_0m20_2018-01-01.jp2. À la ligne Spectral filtered output, nous indiquons un chemin et un nom pour le raster lissé qui sera créé, nous pouvons le nommer place_italie_smooth.tif. Les paramètres suivants peuvent être modifiés ou laissés à leurs valeurs par défaut :
Spatial radius: le rayon maximal (en pixels) sur lequel les zones seront lisséesRange radius: l’écart de radiométrie maximal pour que deux pixels soient lissés
Autrement dit, deux pixels qui seront distants de moins d’un spatial radius et qui auront une différence de radiométrie inférieure à un range radius seront lissés. C’est-à-dire une même valeur moyenne dans chaque bande leur sera attribuée. Par contre, deux pixels éloignés de plus d’un spatial radius ne seront pas lissés. De même que deux pixels dont les radiométries diffèrent de plus d’un range radius ne seront pas lissés, même si ils sont spatialement voisins.
Les autres paramètres peuvent être laissés à leurs valeurs par défaut.
Avertissement
OTB lit sans problème les images au format .jp2 mais peut rencontrer des problèmes pour écrire des images dans ce format. Il est conseillé d’exporter les résultats OTB en .tif.
Le résultat est un raster au format .tif de même résolution et de même emprise que le raster initial mais dont les valeurs ont été localement lissées (Fig. 397). Nous constatons que ce lissage permet de réduire ce qui pourrait s’apparenter à du bruit. Les zones similaires voisines ont été comme aplanies.

Fig. 397 Zoom sur l’orthophotographie initiale (A) et sur la même portion mais sur le raster lissé (B).
Segmentation de l’image (lissée)
Une fois l’image lissée (Lissage de l’image initiale) il est maintenant possible de la segmenter. Le module conseillé se nomme LSMSSegmentation. LSMS signifie Large Scale Meanshift Segmenation. C’est-à-dire que cette segmentation emploie la méthode dite Meanshift (une méthode de segmentation couramment employée) codée pour s’appliquer aux objets à large échelle (aux images lourdes). Le module se paramètre comme présenté sur la figure suivante (Fig. 398).

Fig. 398 Segmentation de l’image précédemment lissée dans OTB.
À la ligne Filtered image nous indiquons l’image à segmenter c’est-à-dire l’image lissée que nous avons précédemment calculée place_italie_smooth.tif. À la ligne Output labeled image nous indiquons un chemin et un nom pour l’image segmentée qui sera produite place_italie_segments.tif. Concernant le Spatial radius et le Range radius, nous pouvons laisser les valeurs par défaut ou bien les changer. Dans notre cas, nous mettons le Range radius à 5. Les autres champs peuvent conserver leurs valeurs par défaut. Les deux champs Size of tiles in pixel (X-axis) et (Y-axis) correspondent à la taille de la fenêtre qui sera utilisée pour accélérer le traitement de l’algorithme. En effet, ce module, pour accélérer les calculs, découpe l’image en sous images, dont la taille est définie par ces deux champs, et segmente ces sous images une par une. Les différentes sous segmentations sont ensuite fusionnées en une seule.
Le résultat est un raster découpé en segments, où chaque segment correspond à une région homogène de l’orthophotographie initiale (Fig. 399). Chaque segment possède un identifiant unique.

Fig. 399 Résultat de la segmentation au format raster.
Note
Ne trouvez-vous pas qu’il y a un petit côté artistique de type tableau fauviste de Derrain ?
Fusion des petits segments
Cette étape est tout à fait facultative mais peut s’avérer très intéressante. Il est possible que lors du processus de segmentation des très petites zones aient été individualisées. Ces petites zones peuvent être constituées d’un seul pixel ou d’une poignée de pixels. Même si ces zones respectent les critères de segmentation définis précédemment, elles ne sont pas forcément pertinentes et vont alourdir fortement le poids du fichier vecteur qui sera créé par la suite.
Il est donc possible de simplifier notre raster de segmentation en fusionnant les zones plus petites qu’un certain seuil avec la zone voisine la plus similaire et de taille supérieure au seuil fixé. Cette opération se fait en utilisant le module SmallRegionsMerging (Fig. 400).
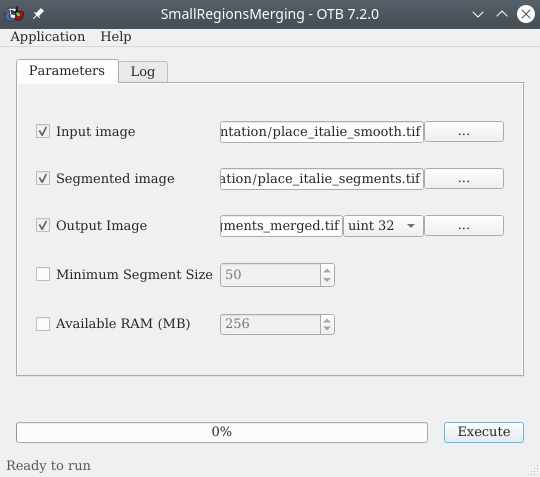
Fig. 400 Fusion des petits segments issus de la segmentation.
À la ligne Input image nous renseignons l’image lissée place_italie_smooth.tif. À la ligne Segmented image, nous vers le raster des segments place_italie_segments.tif. À la ligne Output image, nous indiquons un chemin et un nom vers la couche raster qui contiendra les segments fusionnés, par exemple place_italie_segments_merged.tif. Enfin, à la ligne Minimum Segment Size, nous définissons la taille minimale des segments en pixels, ici nous pouvons laisser 50. Concrètement, tous les segments de moins de 50 pixels seront fusionnés au segment de plus de 50 pixels adjacent et le plus similaire.
Vectorisation de la segmentation
L’étape suivante consiste à vectoriser ce raster de segmentation, éventuellement fusionné. Chaque segment sera ainsi transformé en un polygone de type vecteur. Cette transformation permettra par la suite de calculer des primitives (i.e. des caractéristiques) de chaque segment. Ce processus de vectorisation peut se faire en utilisant un module dédié de OTB LSMSVectorization (Fig. 401). De plus l’intérêt de ce module est qu’il calcule par défaut pour chaque segment les valeurs moyennes des différentes bandes spectrales initiales, ainsi que les variances de ces bandes spectrales pour chaque segment.
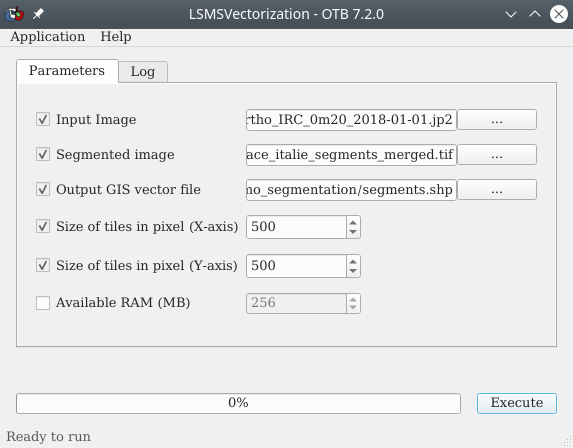
Fig. 401 Vectorisation d’un raster de segmenation avec OTB.
À la ligne Input image, nous indiquons l’image initiale à savoir l’orthophotographie non lissée Place_Italie_ortho_IRC_0m20_2018-01-01.jp2. Cette indication permettra au module de calculer les moyennes et les variances de chaque bande spectrale pour chaque segment. À la ligne Segmented image nous indiquons le raster résultat de la segmentation tel que calculé à l’étape précédente (Segmentation de l’image (lissée)) place_italie_segments.tif ou alors cette segmentation avec les plus petits segments fusionnés place_italie_segments_merged.tif comme expliqué ici : Fusion des petits segments. À la ligne Output GIS vector file, nous indiquons un chemin et un nom pour le fichier vecteur de type polygones qui sera créé place_italie_segments.shp. Les autres paramètres peuvent conserver leurs valeurs par défaut.
Avertissement
OTB ne gère pas encore très bien le format Geopackage. Il est donc plus prudent de travailler au format shapefile. Notez également qu’il est nécessaire d’écrire à la main l’extension .shp dans le nom du fichier exporté pour que le module fonctionne.
Le résultat est un fichier vecteur contenant autant de polygones que de segments issus de la segmentation, ce qui représente souvent un très grand nombre (Fig. 402). Il est possible de superposer ces polygones à l’orthophotographie initiale pour apprécier visuellement le résultat (Fig. 402).

Fig. 402 Polygones des différents segments superposés à l’orthophotographie initiale.
Même si tous les segments identifiés n’apparaissent pas comme étant pertinents nous constatons tout de même que les différents objets de l’image semblent avoir été individualisés. Nous pouvons tout de suite voir que les ombres constituent un problème majeur dans ce type d’approche.
Cette couche de polygones possède une table attributaire dans laquelle chaque ligne correspond à un polygone, i.e. un segment. À chaque segment sont associées différents attributs :
label : un label unique pour chacun des segments.
nbPixels : le nombre de pixels contenus dans le segment.
mean : les colonnes commençant par mean contiennent les valeurs moyennes de chaque bande du raster initial sous chaque segment.
var : les colonnes commençant par var contiennent les variances de chaque bande du raster initial sous chaque segment. De façon logique, les segments ne comportant qu’un seul pixel ont une variance de 0.
Avertissement
Il se peut que la couche vecteur générée présente des erreurs de géométrie (des points redondants, des lignes qui se recoupent …). Si tel est le cas, il est nécessaire de réparer la couche, par exemple en utilisant le module réparer les géométries de QGIS.
Note
L’étape de fusion des petits segments (Fusion des petits segments) permet d’obtenir une couche vecteurs contenant 103400 polygones contre 3523543 pour la version sans fusion. Hormis le fait que la couche générée avec l’option de fusion sera beaucoup plus facile et rapide à manipuler, les segments extraits ne perdent pas forcément en pertinence (Fig. 403).

Fig. 403 Segmentation avec l’option de fusion (A) et sans l’option de fusion (B).
Segmentation de l’image en une seule étape
Comme dit en introduction, si les trois étapes discutées précédemment permettent de bien visualiser chaque sous-étape, OTB propose également un module permettant de chaîner ces trois sous-étapes en un seul traitement. C’est le module LargeScaleMeanShift qui fera ce chaînage (Fig. 404).
Astuce
Dans la phase exploratoire de la méthodologie il est bon de faire les sous-étapes une par une pour visualiser les résultats intermédiaires. Et une fois la méthodologie mise au point, il est possible de l’appliquer de façon plus automatique avec le module permettant de tout chaîner d’un coup.
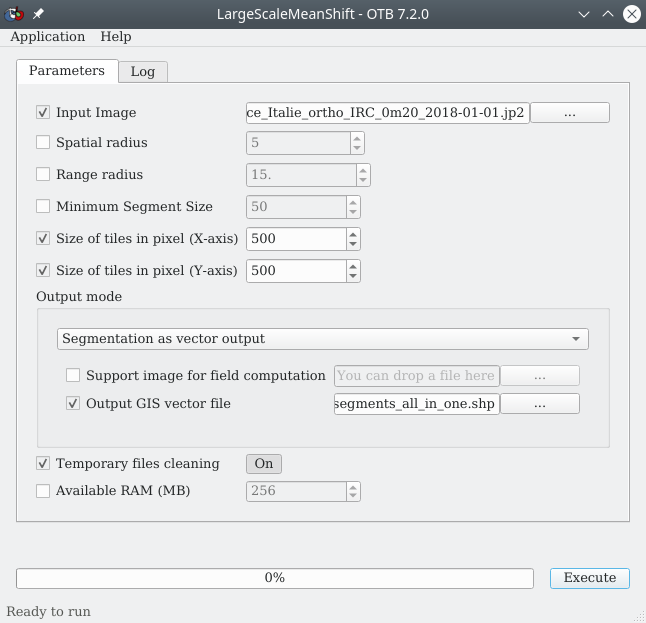
Fig. 404 Segmentation en une seule étape dans OTB.
À la ligne Input image nous indiquons l’orthophotographie initiale Place_Italie_ortho_IRC_0m20_2018-01-01.jp2. Nous pouvons régler ou laisser par défaut les valeurs du Spatial radius et du Range radius. Reportez vous ici pour des détails sur ces deux paramètres : Radius. Dans le panneau Output mode, nous spécifions si nous souhaitons le résultat sous forme de raster ou de vecteurs de types polygones. Ici, nous choisissons une sortie sous forme de vecteurs en sélectionnant Segmentation as vecteur output, puis à la ligne Output GIS vector file nous indiquons le chemin et le nom du fichier vecteur qui sera créé, par exemple place_italie_segments_all_in_one.shp.
Avertissement
Cette méthode All in One produit des résultats quelque peu différents. Les segments sont un peu plus détaillés que ceux obtenus avec les trois sous-étapes effectuées une par une.
Calcul de primitives
Un résultat de segmentation n’est intéressant que pour l’exploitation qui s’ensuit. Une fois une couche de polygones de segments dérivée d’une image initiale, la plupart du temps nous devons classifier les segments de cette couche afin d’en dériver une occupation du sol ou bien d’extraire un type d’objets bien précis. Cette analyse se fait, la plupart du temps, par un processus de classification supervisée. Un tel processus suppose de décrire les polygones de segments en fonction de caractéristiques, autrement dit de primitives, pertinentes. C’est à partir ce ces primitives que le modèle que nous construirons apprendra à reconnaître les différents objets. Par défaut, OTB nous calcule déjà les valeurs moyennes pour chaque bande ainsi que les variances pour chaque bande et chaque segment (Vectorisation de la segmentation). Ainsi, pour chaque segment nous disposons déjà des moyennes et des variances des bandes sous-jacentes.
Si ces primitives de base ne suffisent pas, c’est à l’utilisateur d’en ajouter. Ces primitives peuvent être des caractéristiques associées directement aux polygones comme la superficie, le périmètre ou un indice de forme comme le rapport entre superficie et périmètre. Ce type de primitives s’ajoutent très facilement en calculant ces nouveaux champs pour chaque polygone. Pour plus de détails sur ces manipulations, vous pouvez vous référer au chapitre consacré (Calcul de champs).
Pour les primitives portant sur des valeurs de bandes ou d’indices sous-jacents aux polygones, nous utiliserons le concept de Statistiques zonales pour associer des valeurs moyennes ou de variances (ou tout autre statistique) à chacun des polygones. Ici, par exemple, nous souhaitons extraire la végétation de notre orthophotographie, il est donc pertinent d’associer un NDVI moyen pour chaque polygone. Nous pouvons penser que les segments de végétation auront en effet un NDVI plus élevé que les autres. La première étape consiste donc à calculer le raster de NDVI à partir de notre orthophotographie (Fig. 405). Ce calcul peut se faire via la calculatrice raster de QGIS par exemple (Calcul raster mono-bande).

Fig. 405 NDVI calculé à partir de l’orthophotographie initiale. Les pixels en vert foncé sont ceux à fort NDVI.
Avertissement
Les valeurs radiométriques des pixels d’une orthophotographie ne sont pas exactement comme des réflectances issues d’images satellites, le NDVI est donc légèrement différent, un peu décalé vers les valeurs basses.
Nous pouvons également noter que OTB propose un module pour calculer le NDVI, et d’autres indices, à partir d’un raster multi-bandes, nommé RadiometricIndices.
Hormis les valeurs de bandes et les indices, il peut également être intéressant de calculer des rasters d’indices de texture. Ces indices de texture renseignent sur l’homogénéité, l’hétérogénéité ou la granularité sur une ou plusieurs bandes. Par exemple, sur une orthophotographie les routes ou les toits apparaissent comme étant lisses alors que les espaces arborés apparaissent granuleux (Fig. 397 A). Les indices de texture renseignent de façon quantitative sur ces aspects.
Une fois ces indices radiométriques ou de textures calculés, nous devons les associer aux polygones de segments par statistiques zonales. Pour rappel, il est possible de combiner plusieurs rasters de primitives sous forme d’un Raster virtuel puis de calculer des statistiques zonales sur ce raster multi-bandes pour chaque polygone (Statistiques zonales sur raster multi-bandes avec QGIS). OTB propose également un module de calcul de statistiques zonales nommé ZonalStatistics.
Avertissement
Si la géométrie de la couche présente des erreurs, réparez la à l’aide du module de QGIS Réparer les géométries.
Quelque soit les outils employés, à la fin de cette séquence, nous devons avoir un fichier vecteur de type polygones, où à chaque polygone sont associées différentes valeurs qui peuvent être des statistiques zonales de différents indices radiométriques ou texturaux, des statistiques zonales de bandes ou des caractéristiques relatifs à leurs formes. C’est sur ce fichier vecteur que se fera l’étape suivante à savoir la classification. Dans notre exemple, nous avons ajouté le NDVI moyen et la variance du NDVI pour chaque polygone issu de notre segmentation. Le tout est sauvé dans une couche que nous nommons par exemple segments_primitives.gpkg.
Classification des segments
Une fois les segments caractérisés par l’ajout de primitives, nous pouvons passer à la phase de classification de ces segments. La plupart du temps nous procédons par classification supervisée. Orfeo ToolBox propose un module pour classifier des segments selon plusieurs algorithmes standards comme le Random Forest, le Support Vector Machine, le KNN classifier, …
Comme tout processus de classification supervisée, la première chose à faire est de définir les classes que nous souhaitons obtenir, et de leur associer un identifiant unique. Dans notre exemple, nous souhaitons nous focaliser sur la végétation, nous viserons donc la classification décrite dans le tableau suivant.
ID classe |
Label classe |
|---|---|
1 |
Végétation |
2 |
Bâti |
3 |
Ombres |
Par Bâti nous comprenons tout ce qui n’est pas de la végétation à savoir les routes, les toits, les façades, … La classe Ombres n’est pas une vraie classe d’occupation du sol bien sûr, mais nous sommes souvent obligés d’individualiser les ombres lorsque nous travaillons avec ce type d’images.
Segments d’apprentissage
Ce module nécessite en premier lieu la construction d’une nouvelle couche de polygones qui servira de couche d’apprentissage. Pour créer cette couche, nous commençons par dupliquer la couche de polygones de segments qui contient les primitives segments_primitives.gpkg. Nous pouvons nommer cette couche dupliquée qui nous servira à l’apprentissage segments_train.gpkg. Une fois cette couche dupliquée, nous l’ouvrons dans QGIS par exemple et nous la superposons à l’orthophotographie initiale. Le but est d’identifier visuellement un certain nombre (trois ou plus) de segments représentatifs de nos 3 classes. Pour cela, nous créons deux nouveaux champs dans la table attributaire (Calcul de champs). Nous nommons le premier id_classe et nous le définissons comme étant de type Entier. Ce champ stockera l’identifiant de la classe d’appartenance du segment. Nous créons un second champ nommé label_classe de type Texte qui nous servira simplement à plus facilement identifier les classes par leur vrai nom.
Une fois ces deux champs créés, nous sélectionnons quelques polygones que nous identifions comme étant de la Végétation arborée (classe 1) et nous mettons à jour les champs id_classe et label_classe de ces polygones. Nous procédons de même pour les deux autres classes.
Avertissement
À chaque mise à jour de la table, vérifiez bien que vous ne mettez à jour que les champs des entités sélectionnées !
Une fois ces polygones d’apprentissage renseignés, nous sélectionnons tous les polygones dont les champs id_classes et label_classe ne sont pas renseignés (Requêtes attributaires dans QGIS) et nous les supprimons. Nous disposons ainsi d’une couche ne présentant que des polygones clairement identifiés. Ce sont sur ces polygones que nous allons faire apprendre le modèle de classification.
Segments de validation
Comme pour tout processus de classification supervisé, nous devons associer une phase de validation à notre étude. La validation consistera à comparer les segments classifiés suite à l’apprentissage à certains segments que nous aurons au préalable identifiés pour la validation. Nous devons donc créer une couche de segments de validation en tout point identique à celle que nous avons créée pour les segments d’apprentissage (Segments d’apprentissage). Cette couche ne doit contenir que les polygones renseignés pour la validation. La seule chose à faire attention est de ne pas sélectionner de polygones qui ont déjà servi à l’apprentissage. Les polygones des deux couches d’apprentissage et de validation ne doivent donc pas se superposer du tout. Nous pouvons nommer cette couche de validation segments_valid.gpkg.
À la fin de cette étape nous disposons d’une couche de polygones (de segments) d’apprentissage et d’une couche de polygones (de segments) de validation (Fig. 406).

Fig. 406 Segments d’apprentissage en orange et segments de validation en vert.
Avertissement
Les champs contenant les identifiants numériques des classes doivent être nommés de la même manière dans les couches d’apprentissage et de validation.
Processus de classification
Une fois ces deux couches créées et renseignées, il est temps de passer à l’étape de classification proprement dite. Cette étape peut se faire directement dans Orfeo ToolBox en deux phases :
phase d’apprentissage : nous faisons apprendre à un modèle à reconnaître l’occupation du sol des différents polygones grâce à la couche d’apprentissage (Segments d’apprentissage)
phase d’application du modèle : nous appliquons le modèle construit à tous les polygones non renseignés afin de déterminer à quelle occupation du sol ils appartiennent.
Phase d’apprentissage
Cette phase d’apprentissage se fait à l’aide du module TrainVectorClassifier (Fig. 407).
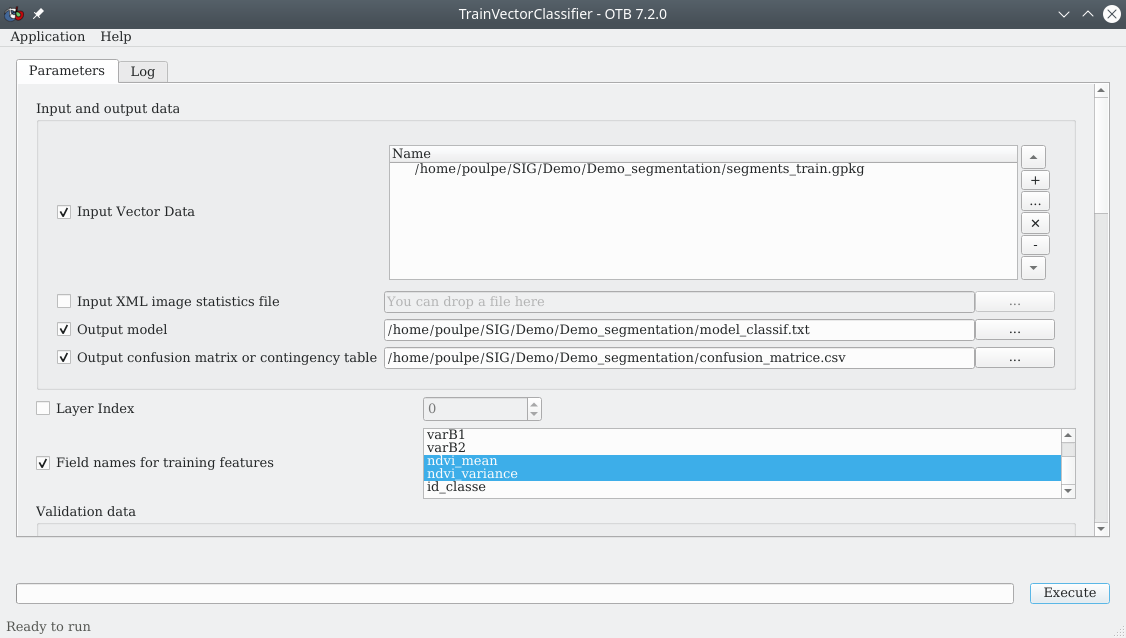
Fig. 407 Entraînement du modèle sur les polygones d’apprentissage.
À la ligne Input Vector Data, nous indiquons la couche d’apprentissage à utiliser, à savoir segments_train.gpkg, via l’icône ![]() . À la ligne
. À la ligne Output model, nous indiquons un chemin et un nom pour le fichier de modèle qui sera créé. Nous pouvons le nommer model_classif.txt. Ce fichier de modèle sera un fichier texte difficilement compréhensible par nous mais néanmoins lisible avec un éditeur de texte. À la ligne Output confusion matrix nous indiquons un nom et un chemin vers la matrice de confusion qui sera créée, par exemple confusion_matrice.csv. Dans le panneau Field names for training features nous indiquons le nom des champs contenant les primitves sur lesquelles le modèle doit apprendre, ici ce sont les deux champs de NDVI ndvi_mean et ndvi_variance.
Dans le panneau Validation data et à la ligne Validation Vector Data, nous spécifions quelle couche de validation nous devons utiliser, ici segments_train.gpkg, via l’icône ![]() . À la ligne
. À la ligne Field containing the class integer label for supervision, nous indiquons quel champ contient les identifiants numériques des classes, à savoir id_classe. Enfin dans le panneau Classifier to use for the training, nous choisissons un algorithme de classification, le Random forests classifier par exemple. Nous pouvons laisser les paramètres de l’algorithme de classification par défaut. Puis nous n’avons plus qu’à cliquer sur Execute.
À ce stade, un simple fichier texte de modèle a été créé.
Application du modèle
Une fois le modèle construit, nous l’appliquons aux segments à classifier à l’aide du module VectorClassifier (Fig. 408).
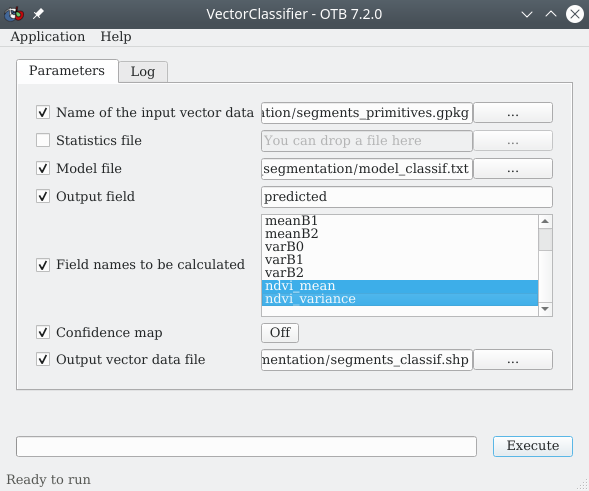
Fig. 408 Application du modèle aux polygones non renseignés.
À la ligne Name of the input vector data, nous sélectionnons la couche des segments à classifier. À savoir la couche vecteur issue de la segmentation qui contient les primitives, segments_primitives.gpkg. Il s’agît de la couche produite à la fin de cette étape : Calcul de primitives. À la ligne Model file, nous pointons vers le fichier texte de modèle généré à l’étape précédente. À la ligne Output field, nous spécifions le nom que prendra le champ qui contiendra les valeurs de classes prédites. Nous pouvons lui laisser sa valeur par défaut.
Dans le panneau Field names to be calculated, nous indiquons les champs qui doivent être calculés lors du processus de classification, à savoir les deux champs de NDVI ndvi_mean et ndvi_variance. Enfin, à la ligne Output vector data file, nous indiquons un chemin et un nom pour le fichier vecteur qui sera créé, par exemple segments_classif.shp. Nous cliquons sur Execute pour lancer le processus.
Avertissement
À ce stade, un bug empêche OTB d’exporter la couche des segments classifiés au format Geopackage, il faut donc l’exporter au format shapefile.
Nous disposons maintenant d’une couche de polygones classifiée selon nos trois classes. Nous pouvons charger cette couche dans QGIS et changer sa symbologie pour apprécier le résultat (Fig. 409).

Fig. 409 Résultat de la classification des segments en vue générale (A) avec la végétation en vert et en vue zoomée (B) avec le détour des polygones de végétation.
Nous constatons que le résultat n’est pas parfait, notamment les arbres qui semblent être les moins en forme ne sont pas repérés, mais dans l’ensemble, le résultat semble satisfaisant.
Validation de la classification
La validation de la classification se fait de façon classique via la matrice de confusion qui a été générée lors de la Phase d’apprentissage. Cette matrice se nomme ici confusion_matrice.csv. Se reporter au chapitre dédié à la lecture des matrices de confusion pour l’interprétation.
Avertissement
Lorsque peu de polygones ont été sélectionnés pour l’apprentissage et la validation, la matrice peut être artificiellement excellente. Dans l’idéal il faudrait sélectionner un grand nombre de polygones surtout pour la validation, et ne pas hésiter à sélectionner des polygones difficiles.