Auteur : Paul Passy
Classification non supervisée d’images satellites
Avant de se lancer dans une classification supervisée de sa zone d’études, il est souvent utile de commencer par une classification non supervisée. Cette approche un peu en aveugle permet de se faire une idée sur la façon dont on pourra différencier les différents usages du sol. Concrètement, l’utilisateur stipule simplement un nombre de classes désirées et laisse la main à l’algorithme pour « faire au mieux ».
Par exemple, si nous visons une classification en 6 classes, nous pourrons tenter une classification non supervisée en 10 classes et faire par la suite un regroupement de classes. Si nous constatons que le résultat est intéressant nous pourrons alors nous lancer dans une classification supervisée avec des réglages plus fins. Par contre, si nous constatons que nous ne parvenons pas à individualiser une classe désirée, peut-être que même avec une approche supervisée nous aurons du mal à la faire ressortir.
L’interprétation des classes sorties automatiquement peut se faire de façon empirique en comparant la classification avec une composition colorée ou une image à plus haute résolution spatiale. Mais l’interprétation peut également se faire à l’aide des signatures spectrales calculées pour chaque classe.
Une classification non supervisée peut être suivie d’un regroupement de classes. Ce regroupement peut se faire avec différents outils.
Ce processus de classification non supervisée peut se reposer sur différents algorithmes mais le plus fréquemment utilisée est celui des K-means (K-moyennes). Cet algorithme peut se mettre en œuvre avec différents logiciels et outils. Certains seront détaillés ci-après.
Pour chacun des exemples, nous ferons une classification non supervisée de l’occupation du sol de la région du Caire en Égypte à partir d’une image Landsat 5 TM du 13 juillet 2011.
Table des matières
Classification non supervisée dans QGIS avec SCP
Version de QGIS : 3.18.2
Version de SCP : 7.8.17
Dans cette partie nous allons voir comment effecteur une classification non supervisée d’images satellites en utilisant le module SCP (Semi-Automatic Classification Plugin) de QGIS. Comme dit dans la partie de présentation des outils, ce module propose de très riches fonctionnalités de classification et de traitements liés à ces classifications. Pour rappel, dans une classification non supervisée, l’utilisateur définit simplement un nombre de classes désiré et un algorithme à utiliser et laisse le logiciel regrouper les pixels en classes de façon automatique.
Avertissement
Le module SCP évolue très vite, de nouvelles mises à jour sont distribuées régulièrement. Lors de ces mises à jour, les fonctionnalités évoluent et l’interface également. La trame général de ce qui est présenté ici ne devrait pas changer mais les détails, notamment les endroits où cliquer et le nom des menus peut évoluer.
Définition du jeu de bandes à classifier
Toute classification s’appuie sur un, ou plus souvent plusieurs rasters à classifier. Ces rasters sont souvent des bandes spectrales mais peuvent également être des indices radiométriques comme des NDVI ou autres. Ici, nous souhaitons classifier les bandes 1, 2, 3, 4, 5 et 7 d’une image Landsat TM afin d’en dériver une occupation du sol. La première chose à faire est de charger ces bandes dans QGIS.
Une fois ces bandes chargées, nous spécifions à SCP que ce sont ces bandes que nous souhaitons classifier. En terminologie SCP, nous allons définir ces bandes comme étant le Jeu de bandes (Band set) à classifier. Pour cela, nous allons dans le menu La fenêtre suivante apparaît (Fig. 336).
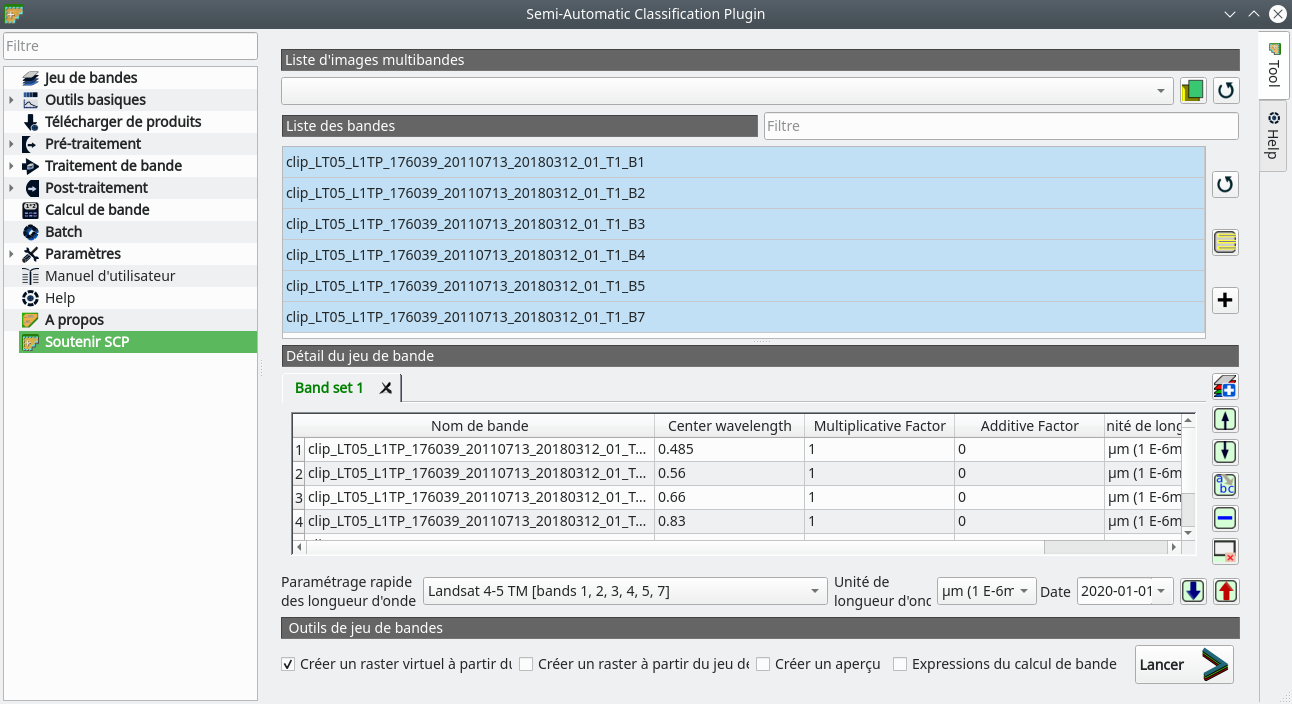
Fig. 336 Définition du jeu de bandes dans SCP.
En premier lieu, nous listons la liste des bandes chargées dans QGIS dans le panneau Liste des bandes en cliquant sur l’icône ![]() . Une fois les bandes listées, nous sélectionnons celles à ajouter à notre jeu de bandes. Ici nous sélectionnons donc toutes nos bandes. Nous les ajoutons à notre jeu de bandes en cliquant sur l’icône
. Une fois les bandes listées, nous sélectionnons celles à ajouter à notre jeu de bandes. Ici nous sélectionnons donc toutes nos bandes. Nous les ajoutons à notre jeu de bandes en cliquant sur l’icône ![]() Les bandes apparaissent maintenant dans le panneau
Les bandes apparaissent maintenant dans le panneau Détail du jeu de bande comme étant le Band set 1. Il est possible de spécifier à SCP que nous travaillons sur des images Landsat 5 TM, en choisissant ce capteur dans le menu déroulant Paramétrage rapide des longueurs d'onde. Grâce à ça, une longueur d’onde centrale est définie pour chaque bande. Il est ensuite possible de créer un raster virtuel à partir ce jeu de bandes en cochant la case Créer un raster virtuel à partir du jeu de bandes dans le bas de la fenêtre. Ensuite, nous cliquons sur Lancer. Nous spécifions un chemin pour la création du raster virtuel et même si nous avons l’impression que rien ne se passe, notre jeu de bandes est créé.
Nous avons maintenant notre jeu de bandes bien défini et un raster virtuel ouvert dans QGIS. À partir de ce raster virtuel, nous pouvons faire une composition colorée en fausses couleurs (PIR, Vert, Bleu) qui va nous permettre de bien identifier visuellement l’occupation du sol.
Paramétrage et classification
Une fois le jeu de bandes défini, la classification proprement dite peut débuter. Pour cela, il est nécessaire d’aller dans le menu . Le menu suivant s’affiche (Fig. 337).
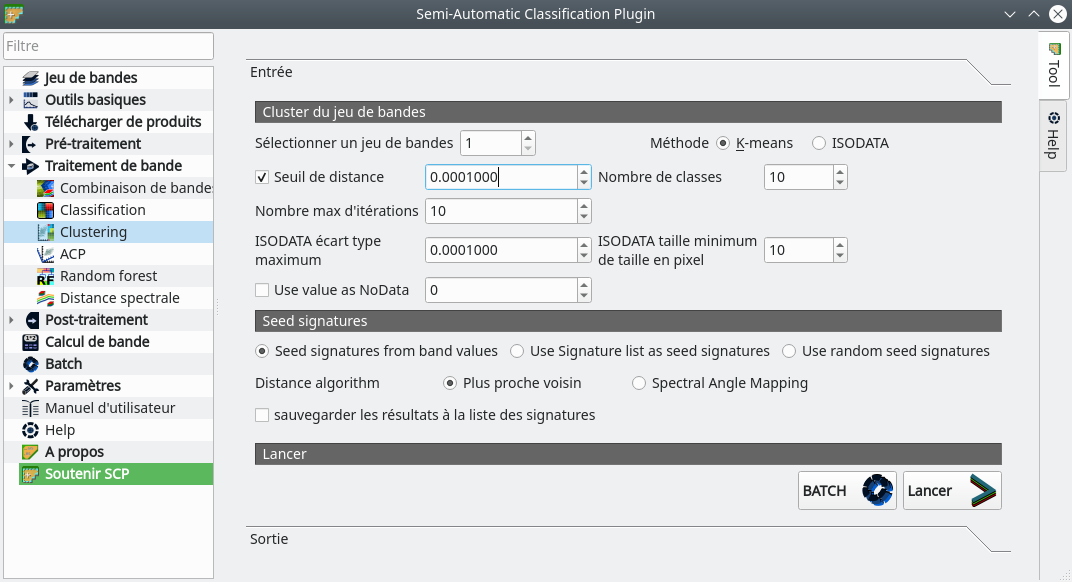
Fig. 337 Définition d’une classification non supervisée dans SCP.
Dans le champ Sélectionner un jeu de bandes, nous indiquons le jeu de bandes à classifier, ici 1. Nous avons le choix entre deux méthodes de classification K-means ou ISODATA. La méthode K-means est la plus fréquemment employée. Dans le champ Nombre de classes nous spécifions le nombre de classes désirées au final, ici 10. Nous pouvons définir un Seuil de distance au dessous duquel l’algorithme de K-means s’arrêtera même si le Nombre max d'itérations n’est pas encore atteint. Les autres paramètres peuvent conserver leurs valeurs par défaut. Il ne reste plus qu’à cliquer sur Lancer. Il est alors nécessaire de définir un répertoire et un nom pour le raster qui sera créé (Fig. 338).

Fig. 338 Classification non supervisée en 10 classes.
Note
Combien de classes choisir ? Au préalable, il est bien de lister les classes d’usages du sol que l’on souhaite obtenir au final. Puis, on gonfle un peu ce nombre afin de voir comment les classes escomptées apparaissent.
En plus du raster classifié, SCP nous fournit les signatures spectrales de chaque classe sous format texte dans la fenêtre de sortie du module (Fig. 339).
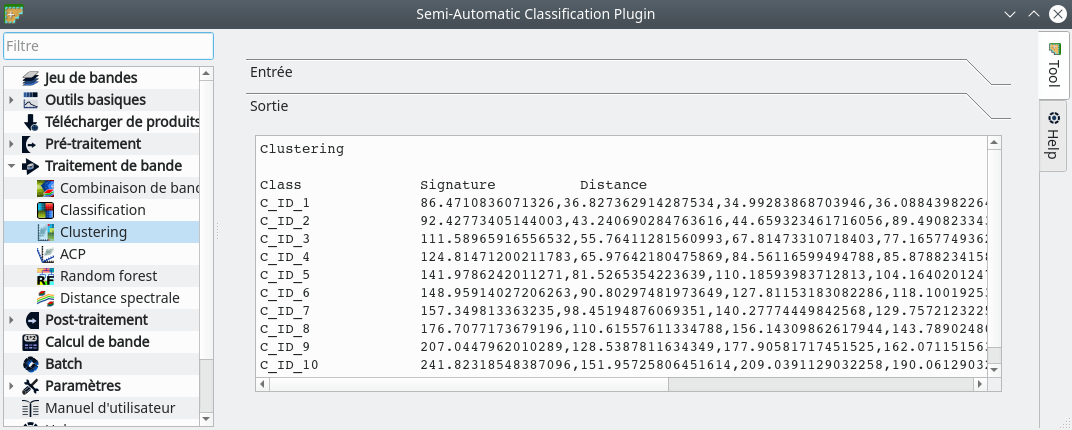
Fig. 339 Signatures spectrales au format texte dans SCP.
Ces signatures spectrales sont également exportées dans un fichier texte dans le même répertoire que le raster. Ce fichier porte le même nom que le raster assorti du suffixe kmeans_report.txt. Dans ce fichier texte, chaque ligne correspond à une classe et chaque colonne à sa valeur moyenne dans chaque bande spectrale du jeu de bandes.
Note
Il est possible d’importer ce fichier de signatures dans un tableur comme Excel ou LibreOffice Calc pour tracer les signatures. Malheureusement, quelques manipulations sont nécessaires pour enlever les lignes inutiles de début et bien régler les séparateurs de colonnes.
Classification non supervisée avec OTB
Version de OTB : 7.2.0
Dans cette partie nous allons voir comment réaliser une classification non supervisée en kmeans en utilisant le logiciel Orfeo ToolBox (OTB). Nous utiliserons OTB au travers de son interface de base Monteverdi. Nous ferons cette démonstration sur la base d’une image Landsat TM prise au dessus du Caire le 13 juillet 2011. Pour rappel, des informations supplémentaires sur les images Landsat TM sont disponibles ici : TM - Landsat 4-5.
Préparation des données
Cette classification se fait sur au moins deux bandes spectrales. Nous construisons donc au préalable un raster multibandes (Raster multi-bandes) contenant seulement les bandes sur lesquelles nous souhaitons baser notre classification. Ici nous la baserons sur les bandes 1 à 6.
Avertissement
Le raster multibandes doit être au format .tif, un raster virtuel ne sera pas géré par OTB comme donnée d’entrée.
Avant de procéder à tout traitement, il est bon de réaliser une composition colorée de notre zone d’étude afin de se rendre compte de sa physionomie (Composition colorée avec QGIS). La figure suivante présente une composition colorée en fausses couleurs de notre image avec le proche infrarouge colorée en rouge (Fig. 340).
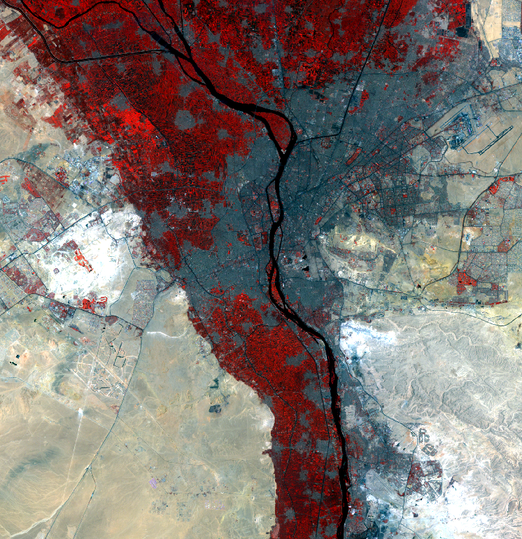
Fig. 340 Composition colorée fausses couleurs du Caire avec la bande du proche infrarouge colorée en rouge.
Sur cette composition colorée (Fig. 340), nous retrouvons les surfaces en eau représentées par le Nil et quelques canaux, du bâti dense au coeur du Caire, du bâti moins dense sur la périphérie de la ville, différents types de végétation notamment à l’entrée du delta et différents types de sols nus aux alentours de la ville.
Maintenant que nous avons un raster multibandes et que nous avons une idée de notre zone d’étude nous pouvons passer à la classification en kmeans dans OTB.
Kmeans dans OTB - Monteverdi
Nous ouvrons Monteverdi qui est l’interface graphique par défaut de OTB. Une fois l’interface ouverte, il est nécessaire d’afficher la liste des applications disponibles en allant dans le menu . Un nouvel onglet nommé Navigateur d’OTB-applications apparaît alors sur la droite. Nous allons utiliser l’application KMeansClassification qui se trouve dans le sous menu Learning. Il suffit d’entrer KMeansClassification dans la barre de recherche pour trouver cette application. Lorsque nous l’ouvrons, le menu suivant apparaît (Fig. 341).
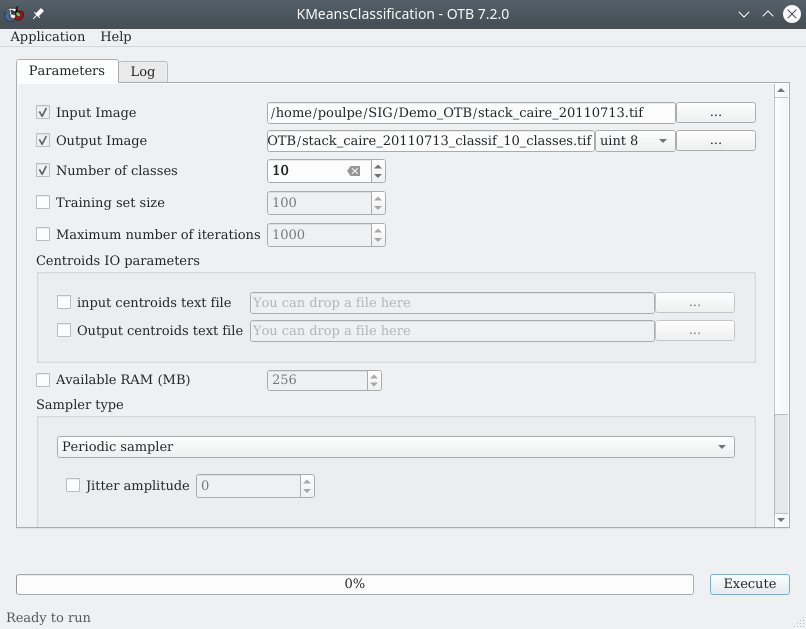
Fig. 341 Paramétrage d’une classification en kmeans avec OTB.
Dans le champ Input Image nous sélectionnons le raster multibandes à classifier, ici stack_caire_20110713.tif. Dans le champ Output Image nous spécifions le répertoire dans lequel nous allons sauver la classification et nous nommons le raster résultant. Dans le champ Number of classes nous spécifions le nombre de classes souhaité, par exemple 10. Plusieurs autres paramètres sont ajustables, mais ces trois points là sont les points essentiels. Nous cliquons ensuite sur Execute.
Le résultat apparaît dans la fenêtre principal de Monteverdi, mais il est possible d’afficher cette classification dans QGIS afin de changer facilement sa symbologie (Fig. 342).

Fig. 342 Classification en 10 classes de l’image Landsat TM de juillet 2011.
Comme pour toutes les classifications non supervisées, le travail suivant consiste à interpréter chaque classe selon sa signature spectrale et à faire des regroupements de classes afin d’obtenir un résultat pertinent. Malheureusement, OTB ne permet pas de calculer facilement les signatures spectrales d’une classification.
Kmeans dans OTB - OTBcli
Il est également possible de procéder à cette même classification mais en passant par la ligne de commande OTB dédiée, comme présentée ci-dessous :
otbcli_KMeansClassification -in stack_caire_20110713.tif -nc 10 -out classif_10.tif
où :
otbcli_KMeansClassification = l’appel de l’application de classification en kmeans
-in stack_caire_20110713.tif = paramétrage du raster multibandes à prendre en entrée
-nc 10 = définition du nombre de classes, ici 10
-out classif_10.tif = le nom de la classification en sortie
Classification non supervisée dans R
Version de R : 4.3.1
Version de terra : 1.7.29
Il est possible de réaliser une classification non supervisée par la méthode des k moyennes (kmeans) dans R en utilisant en partie la librairie terra. Nous commençons par définir un corpus de bandes sur lequel se fera la classification. Il peut s’agir d’une seule bande ou d’un nombre illimité de bandes. Il est également nécessaire de définir un nombre de classes souhaitées, aussi appelé nombre de clusters.
Dans l’exemple ci-dessous, nous réaliserons une classification non supervisée par la méthode des k-moyennes sur une image Landsat 5 (TM - Landsat 4-5) prise au-dessus de la ville du Caire le 13 juillet 2011. Nous nous contenterons des bandes TM suivantes : Bleu, Vert, Rouge, PIR, MIR1 et MIR2. Nous choisirons de créer une classification en 10 classes. Les traitements rasters se feront via la librairie terra mais la classification non supervisée proprement dite sera faite en utilisant la fonction kmeans de r-base.
Nous procéderons en quatre étapes :
Préparation du jeu de rasters à classifier
Classification non supervisée du jeu de rasters
Récupération des signatures spectrales moyennes des classes
Affichage des signatures spectrales moyennes des classes
Préparation du jeu de raster
Nous commençons par importer dans R sous forme de SpatRasters les bandes à classifier puis nous en faisons un stack.
# import des bandes spectrales
b1 <- terra::rast('LT05_L2_176039_20110713_20180312_01_T1_B1.tif')
b2 <- terra::rast('LT05_L2_176039_20110713_20180312_01_T1_B2.tif')
b3 <- terra::rast('LT05_L2_176039_20110713_20180312_01_T1_B3.tif')
b4 <- terra::rast('LT05_L2_176039_20110713_20180312_01_T1_B4.tif')
b5 <- terra::rast('LT05_L2_176039_20110713_20180312_01_T1_B5.tif')
b7 <- terra::rast('LT05_L2_176039_20110713_20180312_01_T1_B7.tif')
# on créé un stack avec ces bandes
stack_Landsat <- c(b1, b2, b3, b4, b5, b7)
# on renomme les bandes de ce stack
names(stack_Landsat) <- c('Bleu', 'Vert', 'Rouge', 'PIR', 'MIR1', 'MIR2')
À cette étape, nous avons un stack de rasters composé de 5 bandes spectrales nommé stack_Landsat. Nous allons pouvoir maintenant lancer la classification non supervisée sur ce stack.
Classification non supervisée
La classification proprement dite se fait à l’aide de la fonction kmeans de r-base. Il faut donc transformer notre stack SpatRaster en une matrice R classique. Nous pourrons ensuite appeler la fonction kmeans sur cette matrice. De façon symétrique, il faudra rappatrier le résultat de la classification généré en matrice dans un objet SpatRaster.
# on transforme ce stack en matrice
stack_mtrx <- as.matrix(stack_Landsat)
# on définit des seeds
set.seed(99)
# on créé les clusters par la méthode des kmeans, en 10 classes et jusqu'à 500 itérations
kmncluster <- kmeans(stack_mtrx, centers=10, iter.max = 500)
# on récupère le résultat de la classification sous forme de matrice
val_cluster <- as.matrix(kmncluster$cluster)
# on créé un raster à 0 à partir d'une des bandes initiales
rast0 <- b1 - b1
# on intègre dans ce raster les résultats de la classification
rast_cluster <- rast0 + val_cluster
# (facultatif) on créé une palette de 10 couleurs
mycolor <- c("#fef65b","#ff0000","#daa520","#0000ff","#0000ff","#00ff00","#cbbeb5","#c3ff5b","#ff7373","#00ff00","#808080")
# on affiche le raster résultat avec la palette
terra::plot(rast_cluster, main = 'Classification non supervisée', col = mycolor, type="classes")
À la fin de cette étape, nous avons un raster avec les 10 classes issues du partitionnement en k-moyennes nommé rast_cluster. Nous y avons associé une palette de couleurs nommée mycolor (Fig. 343). Il est possible d’exporter ce raster si nous le souhaitons (Exporter un raster depuis R).
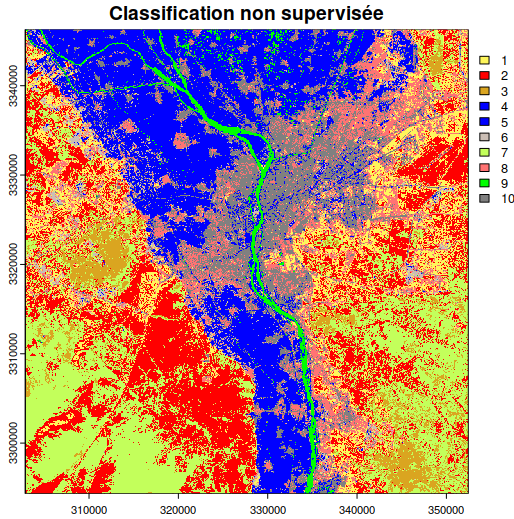
Fig. 343 Classification non supervisée en 10 classes via R.
Récupération des signatures spectrales moyennes des classes
À l’issue d’une classification non supervisée, il est nécessaire d’analyser les signatures radiométriques de chaque classe afin de les interpréter et éventuellement fusionner certaines classes. Nous pouvons facilement récupérer les valeurs moyennes des classes à l’aide de la fonction centers appelée sur un résultat de classification non supervisée générée par la fonction kmeans.
# on créé un vecteur avec les identifiants des classes
id_classes <- c('cl_01', 'cl_02', 'cl_03', 'cl_04', 'cl_05', 'cl_06', 'cl_07', 'cl_08', 'cl_09', 'cl_10')
# on récupère les valeurs moyennes des classes dans un dataframe
sign_classes <- as.data.frame(kmncluster$centers, row.names=id_classes)
Nous obtenons ici un dataframe avec les signatures radiométriques moyennes de chaque classe dans un objet nommé sign_classes. Si nous le souhaitons nous pouvons l’exporter en csv pour l’analyser par ailleurs.
Affichage des signatures spectrales moyennes des classes
Si nous souhaitons rester dans l’environnement R, nous pouvons créer un graphique présentant les signatures spectrales des différentes classes. Nous pouvons utiliser la librairie ggplot2 pour ce faire. Un exemple est donné ci-après. Le but n’étant pas d’apprendre à tracer des graphiques avec R, nous ne nous attarderons pas sur l’explication des différentes options.
# on transforme ce dataframe en format 'long'
sign_classes_long <- tidyr::pivot_longer(sign_classes, cols = c('Bleu', 'Vert', 'Rouge', 'PIR', 'MIR1', 'MIR2'), names_to = "Bande", values_to = "Réflectance")
# on trace le graphique des signatures spectrales moyennes par classe
ggplot(sign_classes_long, aes(x=factor(Bande, level=c('Bleu', 'Vert', 'Rouge', 'PIR', 'MIR1', 'MIR2')),
y = Réflectance,
group = id_cl,
color = id_cl)) +
# Ajout des lignes pour chaque classe
geom_line() +
# Ajout des points pour chaque valeur
geom_point() +
# on associe les mêmes couleurs que pour le raster
scale_color_manual(values = mycolor) +
# Ajout d'un titre général
ggtitle("Signatures spectrales moyennes par classe") +
# on centre le titre et on le met en gras
theme(plot.title = element_text(hjust = 0.5, face = "bold")) +
# on définit un titre pour l'axe des abscisses
xlab("Bande spectrale") +
# on définit un titre pour la légende
labs(color = "Classe")
Il ne reste plus qu’à analyser ce graphique de signatures pour interpréter les différentes classes (Fig. 344).
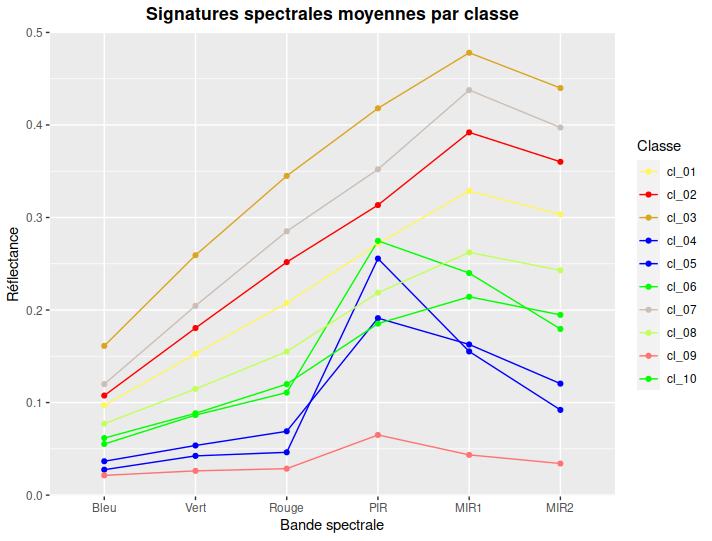
Fig. 344 Signature spectrale moyenne de chaque classe.
Ces signatures sont à mettre en regard du raster produit. Ainsi, la classe 9 présente une signature spectrale très faible dans toutes les gammes de longueurs d’ondes et semble spatialement correspondre au Nil. Nous en déduisons que la classe 9 correspond à de l’eau. Et ainsi de suite pour les différentes classes. Ici, il faudrait certainement fusionner plusieurs classes correspondant à des états de surfaces très proches. Ce type de fusion de classes peut se faire par une reclassification du raster rast_cluster en suivant la méthode présentée dans la partie dédiée (Reclassifier un raster avec R).
Note
Le script complet en un seul tenant est disponible ici : script R kmeans
Cette méthode utilisant R est complète et permet d’automatiser le processus et d’explorer facilement différentes combinaisons de bandes.